So, nachdem wir die letzten Wochen das eine oder andere Mal über Gen-Polymorphismen gesprochen haben, dachte ich, ich mache jetzt auch mal eine ausführliche Gen-Analyse. Ich wurde vorher schon mal auf einzelne Polymorphismen getestet, aber nicht so umfangreich.
Zunächst sollten wir vielleicht klären, was überhaupt ein Gen-Polymorphismus ist. Profis können den Teil des Artikels einfach überspringen.
Das Genetik-1×1
Wie (hoffentlich) jeder weiß, findet sich im Zellkern einer jeden Zelle unsere individuelle Erbinformation – die Gesamtheit dieses Konstrukts nennt sich DNA.
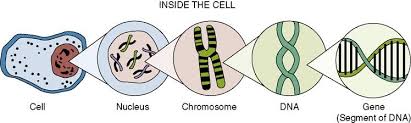
Im Prinzip handelt es sich bei der DNA um ein riesiges Molekül, das Information speichert. Nicht mehr, nicht weniger. Dort finden sich die Baupläne für die unzähligen Proteine im Körper, die chemische Reaktionen ermöglichen.
Die Gesamtheit all der resultierenden chemischen Reaktionen nennen wir dann Biochemie. Die DNA selbst ist sehr lang – die DNA einer einzelnen Zelle wäre ungefähr zwei Meter lang.
Deshalb wird die Struktur gestaucht, wie ein Blatt Papier, das man zusammenknüllt. Das Resultat ist das s. g. Chromosom. Wir bekommen 22 Chromosomen plus 1 Geschlechtschromosom von Mama, und 22 Chromosomen plus 1 Geschlechtschromosom von Papa. Macht unterm Strich 46 Chromosomen.
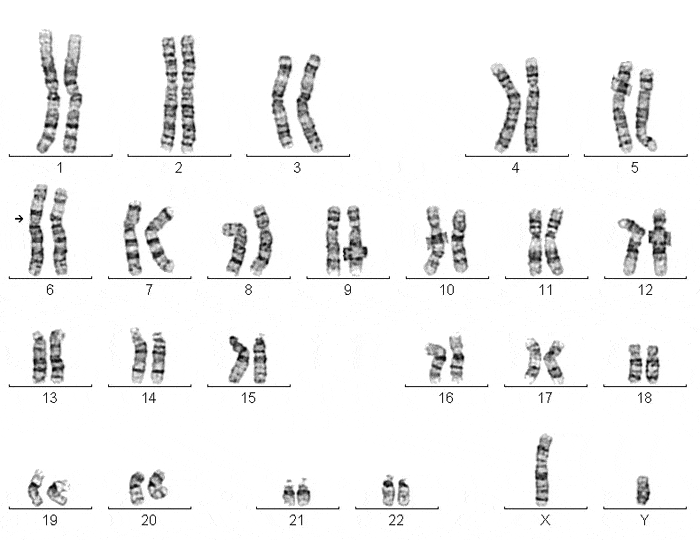
So weit, so gut.
Zoomen wir jetzt wieder zurück in Richtung DNA, finden wir dort einzelne „Informationsabschnitte“, genauer: Baupläne für Proteine, die man Gene nennt. Als Grundlage für das Verständnis der Zellbiologie galt lange die s. g. Ein-Gen-ein-Protein-Hypothese.
Hierbei wird davon ausgegangen, dass ein Gen jeweils den Bauplan für ein Protein trägt. Heute weiß man, dass der Basisgedanke zwar richtig ist, es aber – wie man annehmen kann – weitaus komplexer ist. Ein Gen kann viele Proteine bzw. Proteinvarianten bauen.
Das sind Gen-Polymorphismen
Doch darum soll es heute nicht gehen. Wir wollen über Gen-Polymorphismen sprechen. Denn das ist das, was uns im Einzelnen unterscheidet. Denn die Bananen-DNA gleicht unserer zu 50 %, wir teilen uns 85 % unserer Gene mit der Maus und ca. 60 % der DNA teilen wir uns mit der Fruchtfliege.
Soll heißen: Deine und meine DNA sind zu 99,9 % gleich, und dennoch siehst du vermutlich komplett anders aus als ich (besser so ;-) Späßchen). Häufig sind Gene an genau einer Position verändert – und das sorgt dafür, dass das Protein, was dann gebildet wird, anders ist.
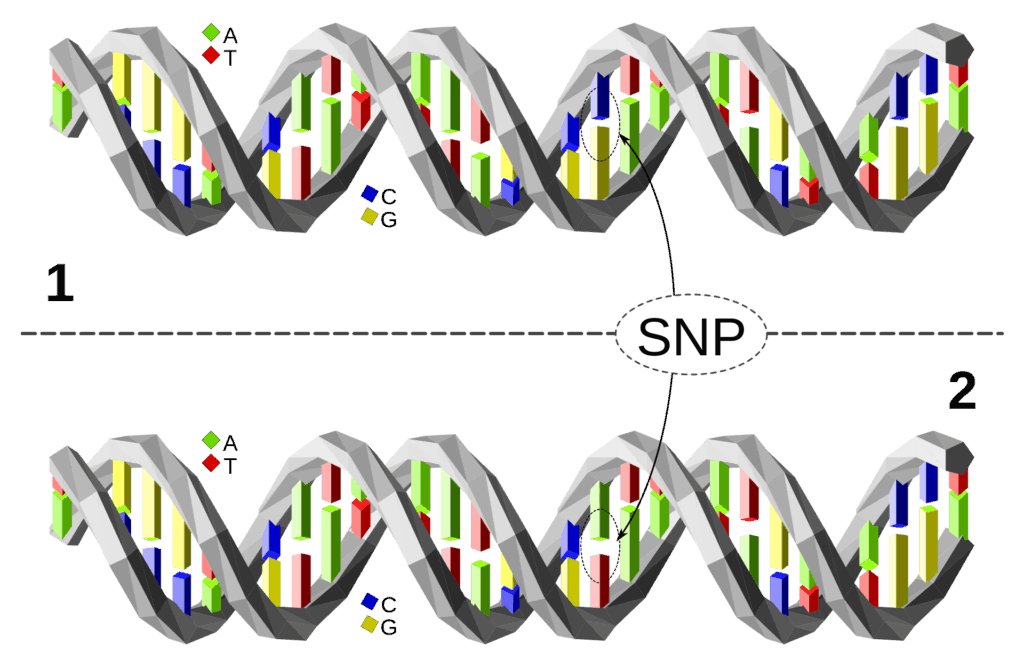
Die Veränderung an genau einer Stelle im Gen nennt sich Single Nucleotide Polymorphism. Die Gesamtheit dieser (marginalen) Veränderungen in all unseren Genen sorgt letztlich dafür, dass wir einzigartig sind.
Wichtig: Die „Sprache“ der DNA besteht aus den vier Basen, die sie in ihrem Aufbau nutzt. Das sind Adenin (A), Guanin (G), Cytosin (C) und Thymin (T) – im Folgenden tauchen immer wieder die einzelnen Buchstaben auf. Das soll nicht verwirren. Man sollte sich nur merken: Wird der Buchstabe getauscht, ändert sich die Information.
Zudem ist im Folgenden häufig die Rede von „Allel“ – ein Gen in Pflanzen kann beispielsweise für die Blütenfarbe codieren. Die Genvariante bestimmt, ob die Blüte die Farbe weiß oder rot trägt. Diese Genvariante nennt man Allel. In diesem Fall gibt es dann ein Allel (= eine Genvariante) für die rote und ein Allel (= eine Genvariante) für die weiße Farbe.
Man kann es auch so ausdrücken: Die Gesamtheit der individuellen Funktionsweise all unserer Proteine im Körper, sorgt für das, was wir sind. Aus dem Genotyp, also der rohen Erbinformation, wird ein Phänotyp, also ein Erscheinungsbild.
Umwelteinflüsse steuern die Gene
Vorab – und das ist wohl das wichtigste Credo dieses Blogs und dem Wissenschaftsfeld der Epigenetik – will ich sagen, dass die DNA im Prinzip etwas in Stein Gemeißeltes ist. Das, was dabei rauskommt, ist es aber nicht.
Denn erst Umwelteinflüsse, die wiederum Einfluss auf die Bildung des jeweiligen Proteins haben, entscheiden über das, was dabei rauskommt – die Rede ist von der Genexpression. Für manche Gene gilt das weniger (z. B. Haut-, Haarfarbe oder Größe), für andere mehr (z. B. wie fit der Muskel ist).
Ein „bekanntes Beispiel“ für die Wirkung von Umwelteinflüssen … sind die eineiigen Zwillinge Otto und Ewald. Der Sage nach war der eine ein Ausdauer- und der andere ein Kraftsportler:

Der Gen-Test – Vorgehen und Analyse
Der „praktische Teil“ der Gen-Analyse ist denkbar einfach. Der von mir gewählte Anbieter 23andme schickt einem ein Kit zu, das wirklich idiotensicher und einfach zu bedienen ist. Es gibt ein Röhrchen, in das man spucken muss. Das ist nicht ganz so easy, wie es klingt, weil recht viel Speichel benötigt wird.
Danach drückt man den speziellen Deckel drauf, in dem sich eine Flüssigkeit befindet, die die DNA-Probe des Speichels konserviert. Man legt das Röhrchen zurück in die Verpackung, und gibt es kostenfrei bei der Post ab. Sechs bis acht Wochen später gibt’s dann die Resultate.
Nette Infos, aber keine Überraschung
So, nachdem die Grundlagen geklärt sind, will ich mal kurz zusammenfassen, was mir meine Gen-Analyse gebracht hat. Nachdem ich circa 50 Stunden damit verbracht habe, dem ganzen Datenpaket einen Sinn zu verleihen, und es durch gefühlt alle Datenbanken der Welt zu jagen, resümiere ich:
Im Westen nichts Neues.
Damit will ich sagen: Jeder, der sich intensivst mit seinem Körper befasst – und das macht man zwangsläufig, wenn man 70 oder 80 Jahre in diesem Körper lebt –, kann mit seiner Analyse entweder komplett verwirrt oder einfach nur bestätigt werden.
Zuerst mal das, was wirklich interessant ist: 23andme hat mir ausgewertet, dass ich zu 99,7 % Europäer bin – jedenfalls wenn die Kalkulation nicht allzu konservativ eingestellt ist. Ich bin also ein echter Gen-Europäer. 23andme hat das sogar so gut gemacht, dass es fast genau meinen aktuellen Lebensort ausmachen konnte – und hat sehr treffend den Ort lokalisiert, wo meine Familie väterlicherseits herkommt, nämlich Schlesien.
Auswertungen auf anderen Plattformen legen mir sogar genau dar, was für ein Mischwesen ich bin – ich bin Franke, Germane, Wikinger und sogar Skythe. Wenn du auch nicht weißt, was das ist: Das war ein Nomadenvolk aus der eurasischen Steppe. Also ich bin querbeet (fast) alles, was Europa zu bieten hat ;-) Muss gerade selbst schmunzeln.
Zudem kann man richtig tief eintauchen in die eigene Gen-Vergangenheit. Ich will da jetzt nicht zu sehr ins Detail gehen, aber eine s. g. Haplogruppen-Analyse kann die Wege rekonstruieren, die „Ur-Mama“ und „Ur-Papa“ in den letzten Zehntausenden von Jahren gegangen sind.
Und man kriegt berechnet, wie viel Prozent Neanderthaler in einem steckt. Zu meiner Enttäuschung haben 92 % der Nutzer mehr Neanderthaler-Gene in sich als ich – ich bin zu unter 2 % Neanderthaler. Typischerweise tragen Europäer so etwa 1 bis 4 % Neanderthaler-Gene in sich – der Neanderthaler war nämlich vor Homo sapiens in Europa, und es fand eine Verpaarung statt.

Wie man bei der Analyse vorgeht
23andme spuckt nur noch Ancestry- und Trait-Reports aus – keine gesundheitsbezogenen Auswertungen. So erfährt man dann zum Beispiel etwas über die Verwandtschaft (siehe oben) oder über die eigene Haut-, Haar-, Augenfarbe und so weiter.

Allerdings hat man auch einen Zugang zu der gesamten Menge an Daten, also Gen-Polymorphismen, die analysiert wurden. Jemand, der sich auskennt, kann dann entweder gezielt nach den jeweiligen Genen bzw. den entsprechenden Gen-Varianten in der 23andme-Datenbank suchen – oder das ganze Datenpaket einfach runterladen. Es gibt viele s. g. Third-party-Anbieter, bei denen man sein Datenpaket hochladen kann, und die einem entsprechend computerbasierte Auswertungen erstellen.
Mein Vorgehen beinhaltete beide Möglichkeiten. Ich habe mir sowohl einzelne Gene und die unzähligen assoziierten Polymorphismen angeschaut und händisch die Pubmed-Suche bedient, als auch quasi alle Dienste, die ich finde konnte, genutzt, um meine Gene analysieren zu lassen. Die Reports und Auswertungen, die dabei erstellt werden, sind manchmal fabelhaft, manchmal einfach nur schlecht.
Zu den „Risiken und Nebenwirkungen“ solcher Auswertungen sage ich im Verlauf noch was.
Polymorphismus-Auswertung am Beispiel FADS2
Das bringt mich zum nächsten Punkt. Es ist sehr interessant zu wissen, mit welcher genetischen und dadurch enzymatischen Ausstattung man daherkommt. So trage ich z. B. ein „Vegetarier-Gen“ (genauer: Genvarianten) im Stoffwechsel der essentiellen Fettsäuren.
In dem Fall heißt das entsprechende Gen FADS2. Die pflanzliche Omega-3-Vorstufe alpha-Linolensäure (ALA) muss verlängert werden, und es müssen mehr Doppelbindungen eingefügt werden, um z. B. EPA zu machen:
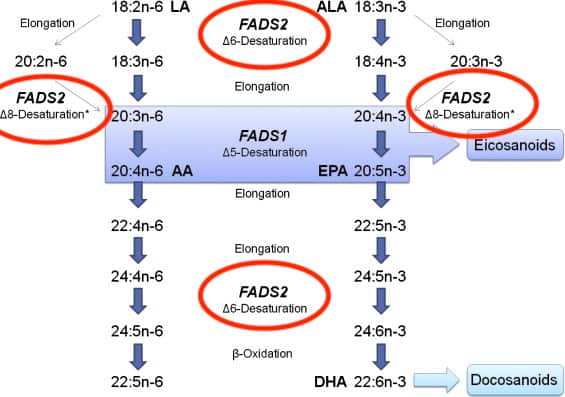
Jeder Gen-Polymorphismus trägt einen spezifischen Namen. Ein einzelnes Gen kann mehrere oder sogar viele Polymorphismen enthalten. Daher versucht die Wissenschaft herauszufinden, welche Polymorphismen im jeweiligen Gen die wichtigsten sind.
In diesem Fall spuckt mir der Report von Dr. Rhonda Patrick bzw. Foundmyfitness den FADS2-Polymorphismus mit dem Namen rs1535(a;a) aus – die Alternative Gen-Variante trägt statt A ein G. Das Gen kann also an dieser Stelle AA, AG, oder GG tragen. Der Report meint:

Diese Gen-Variante also sorgt offensichtlich dafür, dass das gebildete Enzym härter arbeitet. Dadurch kann mein Körper leichter die langkettigen Omega-3-Fettsäuren aus den kürzeren pflanzlichen Vorstufen synthetisieren. Bei den Inuit finden wir das Gegenteil.
So: Wer jetzt aber aufgepasst hat, wird feststellen, dass bei der ganzen Reihe in der obigen Abbildung nicht nur ein Enzym, also FADS2 eine Rolle spielt, sondern auch das Partner-Gen bzw. -Enzym mit dem Namen FADS1. Auch hier habe ich natürlich den einen oder anderen Polymorphismus, der wild mit Daten assoziiert ist:
- Ein Polymorphismus dieses Gens scheint die Aktivität des Enzyms zu senken.
- Der andere Polymorphismus erhöht die Aktivität offenbar.
Schulterzucken.
Meine Erfahrung ist, dass mein DHA- und EPA-Bedarf nicht so hoch ist. Mir reicht es, wenn ich einmal die Woche 200 g Lachs esse – jedenfalls gefühlt. Ich hatte auch nie einen Benefit von zusätzlicher Ergänzung.
Interessante edubily-Polymorphismen
Es gibt ein paar edubily-relevante Polymorphismen in meinem Genom, die spannend sind:
- Ich trage das bekannte „Sprinter-Gen“, die ACTN3-rs1815739(c;c)-Version. Haben 30 % der Europäer.
- In mir scheint HIF1alpha aktiver zu sein – gut für laktazide Sportarten, und gut für die Insulinsensitivität.
- Eine Genvariante macht mir mehr UCP2 (über UCP1 im Fettgewebe sprechen wir des Öfteren) – ein Uncoupling-Protein, das vornehmlich im Muskel vorkommt. Das ist ein natürliches „Antioxidans“, es erhöht den Energieverbrauch und macht warm. Cool.
- Die von mir untersuchten FOXO3A-Genvarianten – auch bekannt als das „Methusalem-Gen“ – haben alle die homozygote Version der nützlichen Allele. Damit soll man u. a. mit bis zu 3 mal höherer Wahrscheinlichkeit weit über 90 Jahre alt werden. Wow ;-)
- Mein Entgiftungsenzym Cyp3A4 arbeitet offensichtlich langsamer. Das verlangsamt den Abbau von bestimmten Medikamenten, einigen Pflanzenstoffen und z. B. Testosteron. Das Enzym findet sich aber z. B. auch im Darm oder auf Immunzellen, wo es für den Abbau der aktiven Vitamin-D-Form Calcitriol zuständig ist – heißt bei mir: höhere lokale Calcitriol-Werte. Bedeutet umgekehrt: Ich brauche ggf. weniger Vitamin D.
- Meine BCMO-Enzyme laufen normal – ich konvertiere gut Vitamin A aus Carotenoid-Vorstufen.
- Ich trage kein „endemisches“ HFE-Gen, das für eine Eisenüberladung sorgen kann. Dafür trage ich die „asiatische“ HFE-Variante, die in ca. 70 % der Asiaten vorkommt, und nur in knapp 10 % bei uns. Das sorgt dafür, dass ich Eisen besser aufnehme. In meinen Augen oft zu viel des Guten.
- Im Homocystein-Stoffwechsel läuft alles tutti bei mir – bis auf die Methionin-Synthase-Reduktase, die ein bisschen langsam ist – sie nutzt B12 als Cofaktor, was bei mir folglich den Bedarf erhöhen sollte. Noch nie was von gemerkt, um ehrlich zu sein.
- Wer jetzt denkt, ich sei der Prototyp eines Landwirts aus dem Neolithikum, weit gefehlt. Ich trage die uralte Jäger-und-Sammler-Variante von CLTCL1 – leider, leider sorgt die alte Variante dafür, dass man schlechter Glukose in Gewebe aufnimmt. Die neue Landwirt- und Farmer-Variante, die während der neolithischen Revolution dominanter wurde, sorgt offensichtlich dafür, dass man Kohlenhydrate bis zum Anschlag schaufeln kann ;-) Trifft nicht auf mich zu.
- Die Gen-Polymorphismen, die ich für die besonders tollen Gene Sirt1, PGC-1alpha und PPARdelta analysiert habe, sind leider leider stinknormal.
Speziell, was sportliche Leistungsfähigkeit angeht, gibt es eine Vielzahl an assoziierten Gen-Polymorphismen. Ich will hier keinen langweilen, deshalb sprechen wir mal noch kurz ein paar andere Themen an. Denn jetzt wird es richtig wild.
Die Glaskugel prophezeit mir Colitis ulcerosa
Zunächst: Das Gute ist, dass ich keine böse Erbkrankheit in mir trage. Jedenfalls die nicht, auf die die vorhandenen Daten getestet wurden. Auf der anderen Seite gehen viele Anbieter jetzt einen Schritt weiter und legen einem Erkrankungsrisiken nahe, indem sie einen s. g. polygenic score berechnen.
Wie der Name vermuten lässt, werden Effektstärken von einzelnen Polymorphismen, die man aus Studien ableitet, addiert, und man bekommt ein Erkrankungsrisiko, das sich aus verschiedenen Polymorphismen zusammensetzt. Hier wird der Tatsache Rechnung getragen, dass nur selten einzelne Gene bzw. Gen-Polymorphismen dafür verantwortlich sind, dass man eine Krankheit bekommt.
Auch hier schneide ich zu meiner Zufriedenheit (oder Beruhigung, je nachdem ;-) ab. „Lustigerweise“ taucht bei einem Anbieter an oberster Stelle das hier auf:

Mein polygenic score, der in diesem Fall angibt, wie hoch mein Risiko ist, im Vergleich zu anderen an Colitis ulcerosa zu erkranken, beträgt über 8. Hinzu kommt, dass ich auf der höchsten Perzentile liege – soll heißen, ich trage ein größeres Risiko als quasi jeder andere Mensch.
Colitis ulcerosa ist eine chronisch-entzündliche Erkrankung des Dickdarms. Der Punkt ist: Mein Dickdarm funktioniert ausgezeichnet. Ich hatte noch nie irgendwelche Dickdarmprobleme – und bin davon überzeugt, auch nie welche zu haben.
Man kann sich in diesen Reports auch genau ansehen, welche Gen-Polymorphismus hier zum Tragen kommen – hier mal eine Auswahl:
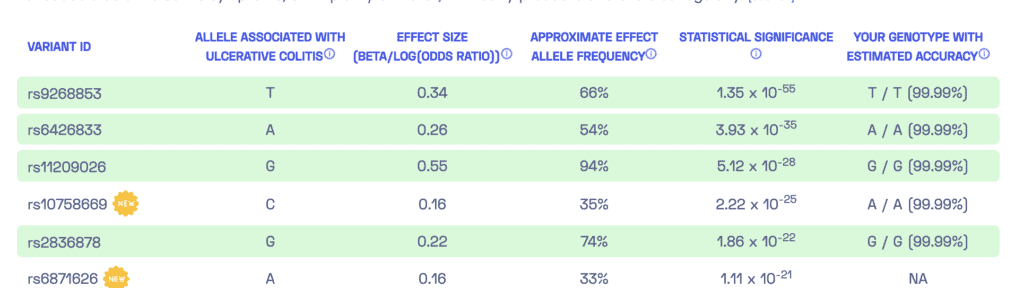
Vorne steht die Bezeichnung, dann das „Risiko-Allel“ – also die Version des Gens, die mit Krankheit assoziiert ist –, die Effektstärke, die Allelfrequenz in der Bevölkerung, die Signifikanz und am Ende der eigene Genotyp. Wer glaubt, das sei ein wildes Harakiri, hat recht. Denn man bekommt schnell den Eindruck, dass das hier höhere Genetiker-Mathematik ist, und dieser polygenic score so etwas wie eine Wetterprognose.
Tatsächlich werden hier Daten aus großen Meta-Analysen abgeleitet – da die Basis dieser Analysen aber noch immer sehr dünn ist, ist die Aussagekraft in den allermeisten Fällen gering. Nur die wenigsten Erkrankungen sind in dieser Hinsicht hinreichend erforscht – und die untersuchten Personengruppen häufig nicht repräsentativ. Man wird mich schwer mit der Petra vergleichen können ;-)
Allgemeine Probleme bei der Interpretation
Ein Laie wird so etwas sehr schnell überfordern. Die dünne Datenlage sorgt zudem dafür, dass wild alles assoziiert wird. Oft wird kein genauer Effekt eines Polymorphismus angegeben (also z. B. schlechtere Enzymfunktion), sondern mit irgendwelchen Endpunkten assoziiert.
Wenn man also glaubt, man würde seine Polymorphismen analysieren und dabei auf eine gut gefüllte Datenlage stoßen, der wird schwer enttäuscht sein. Man kann in den meisten Fällen froh sein, wenn ein Gen-Polymorphismus überhaupt mit irgendwas Sinnvollem assoziiert ist. Das sieht in den meisten Fällen dann so aus:

Wow, wie nützlich. Das heißt: Die meisten Gen-Polymorphismen sind nicht erforscht.
Wie bereits mehrfach angedeutet, kann es ziemlich irreführend sein, einzelne Polymorphismen zu analysieren. Viele Polymorphismen haben einfach keine ausgeprägte Effektstärke. Was interessiert es mich, dass Gen-Polymorphismus X meine Triglyceride (vielleicht, in 5 Jahren, nebenbei, aber auch nur nachts) um 0,2 % erhöht?
Hinzukommt, dass einzelne Polymorphismen im Wirkgefüge einer ganzen Reaktionskette nur eine untergeordnete Rolle haben. Wenn beispielsweise das hypothetisch erste Enzym im Vitamin-D-Stoffwechsel langsamer oder schwächer arbeitet, das hinterste dafür aber besonders gut, wird mir die einzelne Info über die jeweiligen Polymorphismen nichts bringen.
Da es in einem Gen aber unzählig viele Gen-Polymorphismen geben könnte, wir oft aber nur einen Ausschnitt sehen und dann ggf. auch nur den, den wir eben gerade sehen, kann man schnell die falschen Schlüsse ziehen. Hinzu kommt noch, dass man viele Polymorphismen anderer Gene vielleicht noch gar nicht sieht, weil noch nicht erforscht.
Aus genau diesen Gründen kann es sein, dass bei der Auswertung eines Anbieters ein erhöhtes Alzheimer-Risiko bescheinigt wird, während der andere Anbieter einem vorschwärmt, wie toll die eigene Genetik vor Alzheimer schützt. Unterschiedliche Datengrundlage, unterschiedliche Schlussfolgerung.
Es kann auch nützlich und interessant sein
In einigen Fällen, wie zum Beispiel bei OCTN2 (Gen-Name: SLC22A5) – das ist ein Carnitin-Transporter, der dafür sorgt, dass Carnitin in die Zellen kommt –, gibt es durchschlagende Polymorphismen. Bei Verdacht auf Erkrankungen kann man hier eine Gen-Analyse machen, die manchmal bescheinigt, dass das Protein z. B. nicht mehr funktionsfähig ist. In diesem Fall entwickelt sich eine primäre systemische Carnitin-Defizienz, die mit schwerer Stoffwechselentgleisung einhergeht.
Hierfür gibt es spezielle Datenbanken:
Was ich bei der Auswertung sehr spannend fand: Es gibt viele Gene, bei denen kaum Ausreißer auftauchen. Bei diesen Genen trägt man die Allele bzw. die Allelkombinationen, die die meisten Menschen tragen. In vereinzelten Fällen trägt man seltenere Allele. Auf der anderen Seite scheint es Gene oder Gen-Cluster zu geben, die wirklich auffällig sind, wo man sehr viele Allel-Kombinationen trägt, die selten sind:
Spannend wäre, zu wissen, warum solche seltenen Kombinationen gehäuft in bestimmten Bereichen auftreten. Sind sie nützlich? Sind sie gefährlich? Ist es Zufall? Waren sich die Eltern schlicht so ähnlich?
Fakt ist: Ein Nice-to-know für mich war, dass ich einige Risiko-Allele und -Kombinationen in Genen bzw. Gen-Clustern habe, die mit Zöliakie assoziiert sind. Da die Erkrankung einen klaren genetischen Hintergrund hat und sie gut erforscht ist, ist ein möglicher Zusammenhang hier naheliegend. Tatsächlich habe ich auch Gluten-Antikörper. Hier decken sich die Ergebnisse also auch mit meinen Erfahrungen.
Fazit: Was bleibt?
Nun ja. Wer sich die Mühe machen will, seine Daten zu analysieren und daraus einen Sinn zu formen, wird am Ende vermutlich feststellen, dass der Körper in jeder Sekunde des Tages schon genug Sinn aus seinen eigenen Bausteinen gemacht hat. Der Körper macht das so gut, dass es uns in den meisten Fällen gar nicht auffällt.
Soll heißen: Jemand, der ein bisschen mehr Vitamin A braucht, wird vielleicht häufiger zur Leberwurst greifen oder eine Vorliebe für farbiges Gemüse haben. Jemand, der mehr (oder weniger) Omega 3 braucht, wird entsprechend häufiger oder seltener Fisch essen. Und ein Kraftsportler wird in den seltensten Fällen ein 2:30-Marathonläufer.
Die Genetik und Gen-Polymorphismen beweisen uns, dass jeder von uns unterschiedlich und einzigartig ist – sie beweisen uns, dass es kein One size fits all gibt. Sie beweisen uns, dass wir uns mit Blick auf Lebensstil-Ratschläge kaum standardisieren lassen können. Und sie beweisen uns deshalb, dass z. B. Blutwert-Messungen nur dann Sinn ergeben, wenn der Laborbefund sich mit dem eigenen Befinden deckt.
Das, was bleibt, ist das, was wir immer predigen: Das Wichtigste ist, ein Gefühl für den Körper zu kultivieren. Das Körpergefühl bleibt unser wichtigstes Messinstrument. Nur in seltenen Fällen gibt es „unsichtbare Gefahren“, die spezielle Aufmerksamkeit und Handlungsweisen brauchen, und die nicht ohnehin schon durch einen gesunden Lebensstil mit der Beachtung persönlicher Präferenzen abgedeckt sind.
Ach ja, noch ein weises Wort zum Schluss: Gen-Analysen könnten in Zukunft der „Schwanzvergleich“ des 21. Jahrhunderts werden. So auf die Art: „Wow, schau mal, der hat dieses und jenes Gen!“ Auch könnte sich ein Trend ergeben, wobei man sich Partner bewusst nach Gen-Kriterien aussucht. Das wird – wie so oft in der Geschichte des Menschen – in die Hose gehen. Das, was das Gehirn bzw. das Nervensystem und der ganze Körper in Bruchteilen von Sekunden bei echtem Kontakt auswerten kann, werden wir niemals mit solchen banalen Analysen auf einem Blatt Papier hinbekommen.
Daher: Gen-Analysen sind – Stand jetzt – ein nettes, unausgereiftes Tool, das spannende Einblicke liefern aber auch zu Trugschlüssen verleiten kann. In jedem Fall ist zumindest der Deepdive nix für den Laien.

30 comments On Mein Fazit zur Gen-Analyse
Hi! Gibt es mittlerweile nach 5 Jahren neue bessere Anbieter oder Auswerter? Was ist 2025 State of the Art?
Willkommen im Club!
Ich habe das bereits 2015 mal gemacht – seither nutze ich verschiedene Tools – v.a. livewello.com – die sehr ausführliche und immer akutalisierte Reports zu Gesundheit, aber auch Pharmakogenomitk etc anbieten.
Auch Promethease.com ist ein bereits seit einigen Jahren existierendes, sehr detailliertes Sekundärtool.
Allerdings muss man tasächlich sagen, sind die Auswertungen insbesondere in Bezug auf Krankheitsrisiken mit äußerster Vorsicht zu genießen. Selbst wenn ein gering erhöhtes Risiko z.B. von 1,5% bei einer Genvariante X gegenüber Y für eine Krankheit Z angegeben wird, so ist das absolute Risiko immer noch marginal.
Die Stärken sehe ich bisher klar in bereits bekannten und auch in der Labormedizin eingesetzten pharmakogenetischen Merkmalen z.B. zur Verstoffwechselung von bestimmen Medikamenten (schnell, langsam, entsprechend Dosisanpassungen oder Wirkunterschiede möglich) sowie in den oben sehr ausführlich beschriebenen Varianten z.B. bezüglich Sport, Vitaminstoffwechsel etc.
Auch das mit dem Glukosestoffwechsel z.B. ist eine hilfreiche Information, ebenso wie ggf. Salz-Sensitivität oder Eisenspeicherkapazität.
In Kombination mit einer guten Laboranalyse kann man da ja schon einiges für sich herausziehen und ggf. das eine oder andere besser „verstehen“. Denn viele von uns wollen ja gerne besser „verstehen“, warum sich unser Körper in bestimmten Situation oder im Rahmen von Lebensmitteln so oder so verhält.
Vieles, wie z.B. Infos zu sportlichen Fähigkeiten oder Glatzenneigung, wird man im Laufe des Lebens wohl auch selber bereits festgestellt haben und kann sich entsprechen dann „schwarz auf weiß“ in seiner DNA bestätigt sehen. ;-)
Das mit der Verstoffwechselung von Substanzen/Medikamenten unterschreibe ich so zu 100%. Das kann in der (medizinischen) Praxis schon helfen, das stimmt.
23andme bietet doch für 170€ ein „Health + Ancestry Service“
Ansonsten kannst du bei allen deine Rohdna downloaden und auf anderen Seiten analysieren.
Ja, das Kit gibt es, allerdings wird das nicht nach Deutschland geliefert, sondern nur nach Skandinavien und UK. Es gibt die Option „Lieferung in andere Länder“, da ist aber nur das Ancestry Kit verfügbar.
https://www.23andme.com/en-int/
Hier, das. Und dann, wie im Beitrag erklärt, „raw data“ runterladen und woanders hochladen. Es gibt diesbezüglich etliche Tools, z. B. Nebula oder Promethease.
Eben da wird nur noch der „Ancenstry + Traits“-Test angeboten: „Due to applicable regulations, 23andMe only offers an Ancestry + Traits Personal Genetic Service in international markets and health reports are not available“
Auf was willst du hinaus? Das sagen wir doch die ganze Zeit. Es gibt keine Tests mit Gesundheitsaussagen mehr, deshalb muss man diesen Test machen und die Rohdaten runterladen und bei anderen Anbietern hochladen.
Tatsächlich verspricht der „TellmeGen“ DNA Test Aussagen über die Gesundheit und wird auf Amazon.de verkauft:
Chris, wie viel hast du ca. zusätzlich gezahlt für die verschiedenen zusätzlichen Analysen? Das würde mir zumindest dabei helfen abzuwägen, ob die 129€ für TellmeGen, oder „AncestryDNA + viele andere Services die ggf. kosten“ bzw. „23andme + viele andere Services die ggf. kosten“ mehr Sinn macht.
Ich würde mal schauen, welche Aussagen da genau getroffen werden. Ich bezweifle, dass hier aussagekräftige und ausführliche Reports erstellt werden.
Ich denke, alle Dienste, die ich zusammen genutzt habe, haben mich so um die 1000 Euro gekostet. Sehr viel Geld – ich weiß nicht, ob ich das so nochmal machen würde.
Du solltest vorher unbedingt wissen, nach was genau du gucken willst. Entsprechend kommt man auch mit einem Bruchteil an Geld davon.
Angeblich trifft TellmeGen über all dies Aussagen: https://images-na.ssl-images-amazon.com/images/I/71pEOVdTVIL._AC_SL1200_.jpg (und https://images-na.ssl-images-amazon.com/images/I/71ILiA%2BQdlL._AC_SL1200_.jpg bzw. https://images-na.ssl-images-amazon.com/images/I/71KyC5pspSL._AC_SL1200_.jpg)
Oh, krass. Das ist echt eine ganze Menge.
Ich dachte, dass der Umfang der analysierten Gene ein anderer ist, wenn man nur den „ancestry and traits“-Test macht. Oder werden die gleichen Gene analysiert und nur die Auswertung nicht?
Deshalb macht man den doch bei 23andme – weil man jedenfalls von dem Anbieter weiß, dass man die komplette Bandbreite an Genen kriegt, die man dann bei anderen Anbietern analysieren lassen kann. Deshalb sprechen wir doch laufend von „raw data“.
Ich weiß jetzt nicht wie du „komplette Bandbreite“ definierst aber zur Information alles Interesssenten an DNA Analyse:
23andme und Acestry messen ca. 640000-660000 SNPs, ca. 0,2 % des Genoms.
Nähere Infos zu genaueren Angaben der speziell gemessenen SNPs gibt es hier zu sehen.
https://geneticgenie.org/article/best-dna-raw-data-23andme-ancestrydna-or-myheritage/
Stell Dir vor, Du kannst sogar Dein komplettes Genom sequenzieren lassen ;-) Davon kannst Du genau 1 % gebrauchen, die für Proteine codieren. 23andme und Co. liefern einen für Leser dieses Blogs (Laien und sogar Profis) gute und erste Einblicke. Darüber hinaus ist die raw data-file kompatibel mit einer Vielzahl an Third-party-Anbietern. Ich sehe zum aktuellen Zeitpunkt keinen Grund, andere, noch viel unausgereiftere Technologien zu diskutieren.
Das „komplette Bandbreite“ bezog sich auf die Frage, ob die Testart bei 23andme Einfluss auf die Zahl an SNPs hat, die man analysiert bekommt.
Das mit dem ganzen Genom analysieren weiß ich schon ;)
Den Teil mit „Ich sehe zum aktuellen Zeitpunkt keinen Grund, andere, noch viel unausgereiftere Technologien zu diskutieren“ verstehe ich jetzt nicht.
Mit dem Link wollte ich darauf hinweisen das die Anbieter teilweise unterschiedliche SNPs messen. Falls jemand Interesse an bestimmten SNPs hat kann derjenige so besser eine Entscheidung treffen zu welchem Anbieter er geht.
Bsp. 23andme misst mehr mitochondriale SNPs als andere und Ancestry misst mehr SNPs bezüglich Mediakenten.
Aus dem Bericht geht eh hervor das man bezüglich Gesundheits-SNPs eh nur 23andme oder Ancestry wählen sollte.
Wie gesagt, ich finde nicht, dass es eine Relevanz für die Leser dieses Blogs (oder allgemein Leuten, die meistens ein Laien-Verständnis haben und trotzdem ihre Gene analysieren lassen wollen) hat, zu unterscheiden ob man mehr von diesen oder jenen SNPs haben will, weil man im Endeffekt eh nur mit den wenigsten SNPs wirklich was anfangen kann und zudem viele relevanten Gene von allen Anbietern untersucht werden. Und ich sagte, dass man mittlerweile sogar sein ganzes Genom analysieren lassen kann – da wäre der Unterschied deutlich stärker, aber das ist eben unausgereift und bringt uns noch viel weniger unterm Strich.
Hallo,
ist „lustig“, dass der Beitrag ausgerechnet jetzt erscheint, da ich seit etwa 2 Wochen am Überlegen war, spaßeshalber auch eine Genanalyse durchführen zu lassen. Allerdings habe ich bei den ganzen Anbietern nur noch die Abstammungsanalysen gefunden, die Gesundheitsanalysen sind in Deutschland offenbar nicht mehr erhältlich. Auch auf den US-amerikanischen Originalanbieter-Seiten besteht keine Möglichkeit zum Versand nach Deutschland … oder habe ich einfach nur die richtigen Quellen nicht gefunden?
VG und einen schönen Tag an alle!
Hallo Chris,
ich sehe viele Auswertungen auch kritisch, da in der Tat häufig nur Wahrscheinlichkeiten benannt werden. Und die Auswertungen von 24Genetics (wo ich den Test habe machen lassen) fand ich auch nur bedingt brauchbar, wobei die Auswertung zum Thema Sport schon sehr aussagekräftig war..d.h. mich gut beschrieben hat. Aber es gibt doch auch SNPs, die extrem hilfreich sind, wenn man die von sich kennt: wie MTHFR, PEMT oder die GST-Familie. Oder siehst Du das anders?
LG,
Robert
Hey Robert,
ich weiß nicht.
Bei PEMT habe ich z. B. rs7946(C;T), assoziiert mit weniger Phosphatidylcholine. Zeitgleich habe ich aber auch rs174548(C;C) im FADS1-Gen, was mit höheren PC-Werten assoziiert ist. Unterm Strich macht das für mich: keine Real-Life-Relevanz. Ich habe noch nie von Cholin profitiert, weder als -Bitartrat noch als PC direkt. Ich esse ja auch genug, so ist es nicht.
Ich hab zwar ein normales MTHFR, aber den rs1801394(g;g) bei MTRR – das sollte mir hohe Homocystein-Werte bescheren, meinen B12-Bedarf erhöhen und mich zu einem genetischen Hypomethylierer machen. Nichts von dem trifft zu. Ich hatte in Zeiten, wo ich wirklich überhaupt keine Sorge um Ernährung hatte keinen Hcy-Wert über 10. Das ist zwar nicht „Optimum“, aber auch nicht allzu hoch. Ich habe noch nie von B12 profitiert, eher im Gegenteil. Wenn ich meine Methylierungs-Vitamine einnehme, geht es mir eher schlechter.
Gleiches gilt für rs2060793(a;a), Polymorphismus im ersten Enzym vom D-Stoffwechsel – sollte mir niedrigere Calcidiol-Werte machen. Habe ich noch nie was von gemerkt. Ich profitiere auch nicht von Extra-Vitamin-D, im Gegenteil. Wenn ich hochdosiert D einnehme, verschlimmern sich Entzündungen. Wie im Artikel dargelegt: Ich führe das auf andere PM zurück, die eine höhere Calcitriol-Bildung zulassen.
Ich will jetzt nicht sagen, dass ich der tolle Über-Experte bin. Aber wenn es mir schon wenig bringt, was soll dann ein Laie auf dem Gebiet damit anfangen? Ich kann stundenlang durch Promethease scrollen – „assoziiert mit niedrigem HDL“, „assoziiert mit hohem HDL“, „assoziiert mit niedrigem HDL“, … und so weiter. Mein HDL war ohne Anstrengung immer um die 70. Und selbst wenn er das nicht gewesen wäre: Was hätte ich tun sollen? Mir irgendwelche NEM reinziehen, die mir an anderer Stelle das System verziehen?
Das ist auch mein großer (und immer größer werdender) Kritikpunkt an „Blutwert-Optimierung“. Man kommt sich vor wie eine Katze, die vergeblich die Maus jagt. Die kleine Maus versteckt sich irgendwo und taucht plötzlich wieder hinter der Katze auf. Die Katze springt dieser Maus hinterher, nur um irgendwann zu verstehen, dass da nicht nur eine Maus ist, sondern viele Mäuse. Was ich damit sagen will: Das ist eine Never-ending-Story, das ist für mich einfach kein adäquater Zugang, schon gar nicht für jemand, der sich nicht auskennt.
Ich finde, da geht auch der Fokus verloren bzw. wird aufs Falsche gerichtet. Die Leute benutzen immer mehr ihren Kopf, statt ein Körpergefühl zu kultivieren. Wenn mein Hund zu viel von etwas hat, dann riecht die dran, dreht sich um und geht weg. Wir Menschen verlernen das, verlernen die Zugänge zum wichtigsten Messinstrument, wenn wir uns zu sehr auf Zahlen auf einem Blatt Papier fokussieren. Solche Werkzeuge sind nett und helfen auch an der einen oder anderen Stelle, und sicher ist so eine Zahlen- und Datenspielerei für uns Kopfmenschen, Wissenschaftler und kleine „Autisten“ spannend. Aber in meinen Augen steht der Aufwand oft überhaupt nicht in Relation zum Ertrag.
Hallo Chris,
ok, wie Du bei mir im Buch ja gelesen hast, habe ich von einigen SNPs mehr profitiert. Ich habe PEMT aber auch in der Ausprägung T,T, was anscheinend mehr Mangel verursacht, vor allem wenn man eine LCHF/Paleo-Ernährung mit reichlich Sport kombiniert. Ich habe diese Erfahrung ja ausgeführt…und das ist durch die Einnahme von PC weg gegangen (siehe Blutwert „CK“). Das hätte zum echten Problem werden können…meine Schwester hat Ihre Galle zB draußen.
Ich finde aber auch die Auswertung von MAOA und COMT spannend, da die bei mir auch zu 100% zutreffen. Da kann man jedoch weniger direkt dran drehen, aber man weiß nun damit umzugehen.
Ich denke, es ist insgesamt eine gute Mischung aus „korrekten Ernährung“, Bewegung, Blutwerte prüfen (Optimieren hat sicherlich seine Grenzen) und korrigieren wenn echt schlecht, Bauchgefühl …und auf ein paar SNPs achten…wie zB dann auch noch die APO-E4 Geschichte und gesättigte Fette. Ist für E4-ler nach aktuellem Stand gefährlicher oder bedenklicher als für E3/E2-ler. Und da kann man am Ende des Tages immer ein bißchen Risiko aus dem Ballon lassen.
Körpergefühl ist aber ein sehr gutes Punkt, da der uns generell ziemlich verloren gegangen ist. Wir haben kaum noch eine Verbindung zur Umwelt…zum. wenn man in einer Stadt oder Großstadt lebt. Leider.
LG,
Robert
Nochwas…was ich ja im Buch auch anfange, aber wirklich nur anfange ist die Kombination der SNPs miteinander. Wie Du schreibst, MTHFR ist gut, MTRR aber schlecht…SOD2 ist schlecht, GPX jedoch gut…oder eben auch schlecht. Oder die ganzen Permutationen mit Phase1 und Phase 2 Entgiftungsenzymen…
Das ist nochmal eine weitere Dimension, die auch in den ganzen Studien wenig beleuchtet wird…die aber immens wichtig ist. Macht das ganze leider nicht weniger komplex :-D
LG,
Robert
Interessant, die Polymorphismen zum Carnitin-Transporter! Das Thema hatten wir schon mal „bequatscht“ nach einem Newsletter. Bei mir schlägt L-Carnitin richtig gut an (HRS u. Ausdauer).
Welche Art von Stoffwechselentgleisungen hast Du da im Sinn? LDLHDL bzw. Triglyceride?
Bei den „pathogenen“ Carnitin-Mutationen sprechen wir von sehr schweren Entgleisungen, zB Cardiomyopathie, Unterzuckerung und keine Toleranz gegenüber Nahrungsknappheit… das kann ganz schwere Störungen hervorrufen.
Ich habe allerdings einen für Europäer relativ gängigen PM, der die Promotor-Region vom Carnitin-Transporter-Gen verändert – diese PMs gelten als Anpassung im Zuge der neolithischen Revolution. Man weiß nicht so genau, warum es diese PMs bei OCTN1 und OCTN2 gibt, aber man vermutet Anpassungen an Landwirtschaft. Diese nicht-pathogenen Carnitin-PMs stehen aber im engen Zusammenhang mit den bei uns häufig auftretenden Darmerkrankungrn. Es wird vermutet, dass die Darmzellen nicht genug Carnitin abbekommen (aber speziell Darmzellen leben von Fettsäuren), und sich daher das Erkrankungsrisiko erhöht. Es ist also davon auszugehen, dass meine Carnitin-PMs dazu führen, dass ich mehr Carnitim aus der Nahrung brauche, da meine Speicher ansonsten rasch verarmen. Unterm Strich kann man sagen: Es gibt einen Grund, warum die neolithische Revolution solche PMs hervorgebracht hat. Allerdings „funktionieren“ solche PMs nur unter bestimmten Bedingungen … und erhöhen wie erwähnt möglicherweise Erkrankungsrisiken. Wir haben uns die Landwirtschaft also teuer erkauft.
Mal wieder ein sehr aufschlussreicher Artikel. Da es solche DNA Tests ja auch für Sport & Ernährung gibt, wäre das vielleicht eine passende Fortsetzung? In jedem Fall ein äußerst spannendes Gebiet was in der Zukunft mit Sicherheit noch populärer werden wird. Natürlich nur, wenn die Tests und deren Auswertung präziser werden.
Danke dir!
Hätte ich Thema Sport und Ernährung für erwähnenswert gehalten – denn natürlich habe ich meine Gene auch dahingehend analysieren lassen -, hätte ich ausführlich berichtet. :-)
@Chris Michalk
Hat es einen bestimmten Grund wieso du dich für 23andme entschieden hast? Ich kann mich erinnern vor kurzem einen Artikel gelesen zu haben das AncestryDNA mehr gesundheitsbezogene Daten liefert. Mit Ausnahme das 23andme mehr Gene in Bezug auf die Mitochondrien misst.
Bauchentscheidung. 23andme kenne ich Ewigkeiten und ich hatte da irgendwie das größte Vertrauen in die Erfahrung des Unternehmens. Denke aber, dass sich das zwischen den beiden nicht viel schenkt.
Bezüglich der Neandertaler:
Wieso sind 203 von 7462 weniger als 2 Prozent?
Ich gehe davon aus, dass die Berechnung ein bisschen komplexer ist.